Dez anos de neurociência no Google produzem mapas do cérebro humano
O cérebro humano não utiliza mais energia do que uma fraca lâmpada incandescente, mas pode realizar proezas ainda impossíveis com os maiores sistemas de computação artificial. Compreender como é necessário um nível de compreensão mais profundo do que saber que parte do cérebro é responsável por qual função.
O campo da conectômica visa conseguir isso mapeando com precisão como cada célula está conectada a outras. Construir mapas detalhados de como os cérebros de muitos tipos de organismos estão conectados está transformando nossa compreensão de como os cérebros funcionam. Isso poderia ajudar os pesquisadores a compreender os distúrbios neurológicos e também a responder questões fundamentais, como a forma como as memórias se formam.
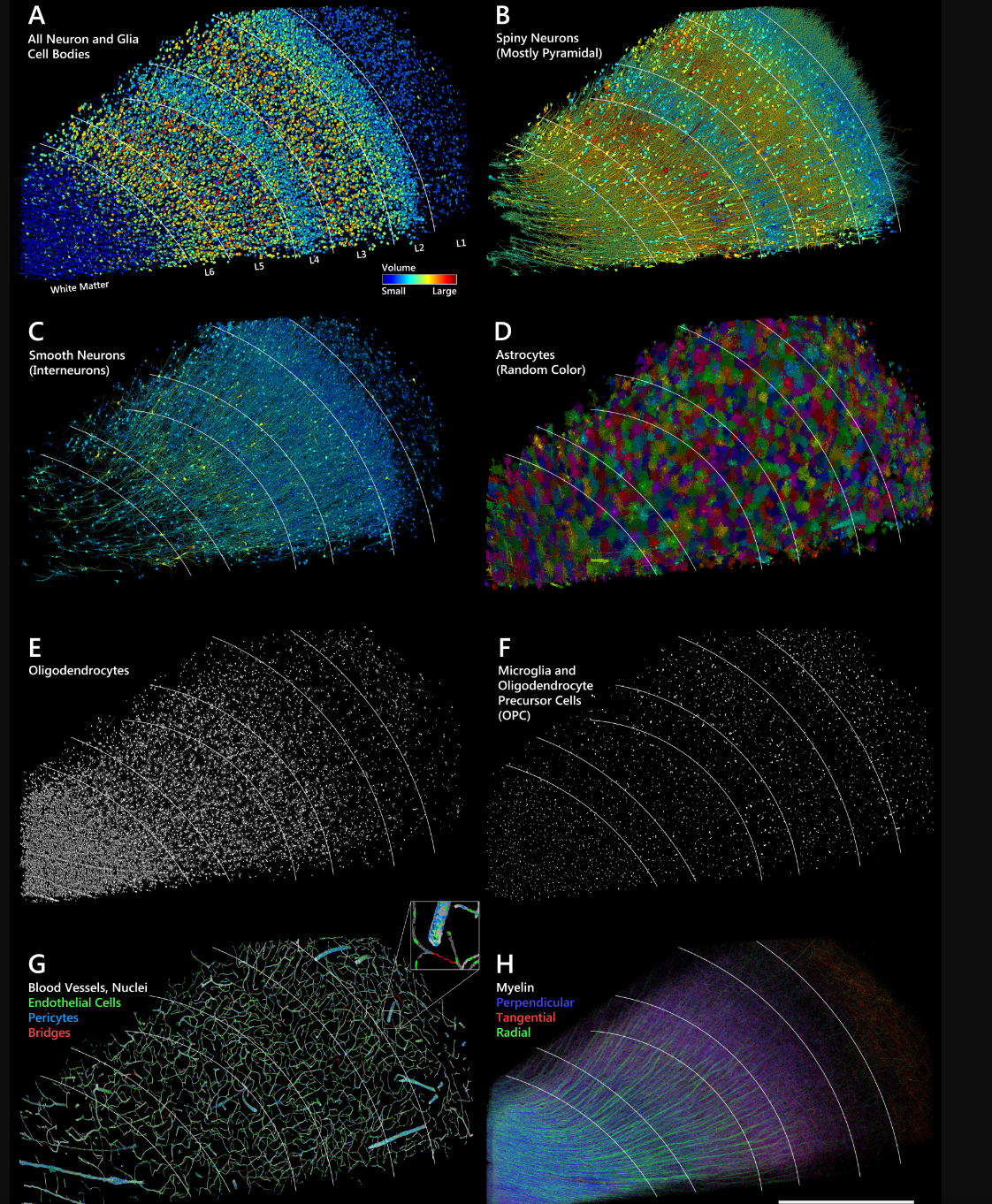
Este ano marca o décimo aniversário da formação da equipe de pesquisa Connectomics do Google Research. Tornamos os conectomas possíveis por meio do desenvolvimento de algoritmos de aprendizado de máquina (ML) e ferramentas de software que processam e visualizam dados em uma escala sem precedentes. Marcando este aniversário, publicamos hoje na Science , “ Um fragmento de petavoxel do córtex cerebral humano reconstruído em resolução em nanoescala ” em colaboração com Jeff Lichtman da Universidade de Harvard e outros.
Lançado anteriormente como uma pré-impressão , este conectoma descreve um pedaço de tecido cerebral humano do tamanho de meio grão de arroz que, no entanto, requer 1,4 petabytes (1,4 milhão de gigabytes) para ser codificado — incluindo cerca de 16.000 neurônios, 32.000 glia, 8.000 células de vasos sanguíneos (para um total de aproximadamente 57.000 células) e 150 milhões de sinapses. Este projeto revelou estruturas nunca antes vistas no cérebro humano que podem mudar a nossa compreensão de como o nosso cérebro funciona.
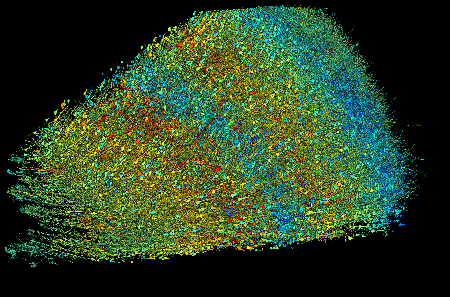
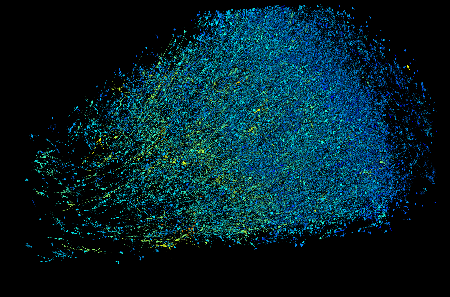
Desde que publicamos o preprint, expandimos nosso conjunto de ferramentas interativas e de código aberto que permitem aos pesquisadores investigar o conjunto de dados por conta própria. A capacidade de outros pesquisadores revisarem e refinarem esse conectoma do cérebro humano é uma das muitas maneiras pelas quais vemos o lançamento deste artigo e das ferramentas associadas não apenas como o culminar de 10 anos de trabalho, mas como o início de algo novo.
Dimensionando a ciência do cérebro
O primeiro conectoma foi publicado em 1986 — antes da existência de ferramentas de IA — para os 302 neurônios do organismo modelo de nematóides Caenorhabditis elegans . Demorou 16 anos para criá-lo a partir de imagens microscópicas transversais do verme. Os pesquisadores coloriram manualmente as células de uma seção transversal para outra para visualizar as conexões neste sistema nervoso simples.
Quando a equipe Connectomics do Google foi lançada, há dez anos, estávamos entusiasmados com a forma como as inovações em IA e o trabalho com grandes conjuntos de dados poderiam nos permitir passar de 302 neurônios para dezenas de milhares ou milhões encontrados em organismos mais complexos. Nosso trabalho exigiu novos algoritmos capazes de lidar com as enormes quantidades de dados — agora petabytes — que esses estudos geram. Desenvolvemos redes de preenchimento para substituir o esforço manual de colorir células em imagens. Estas redes permitem a reconstrução automatizada de neurônios através de camadas de tecido. Com base nisso, nosso algoritmo SegCLR identifica automaticamente partes distintas de células e tipos de células dentro dessas redes. Também desenvolvemos o TensorStore , uma biblioteca de software C++ e Python de código aberto para armazenar e gerenciar enormes conjuntos de dados multidimensionais. Essa ferramenta obteve benefícios muito além da conectômica e agora é amplamente usada no Google e em toda a comunidade de ML mais ampla.
Demonstramos esses algoritmos pela primeira vez quando lançamos o conectoma para o “hemicérebro” da mosca da fruta Drosophila melanogaster em 2020. Revelando as conexões entre 25.000 neurônios em uma porção central do cérebro da mosca da fruta, esta reconstrução foi usada por outros pesquisadores para fazer descobertas sobre aprendizagem, memória e comportamento na mosca da fruta. Desde então, grupos publicaram centenas de artigos baseados no conectoma da mosca-das-frutas.
Através de colaborações com pesquisadores do Howard Hughes Medical Institute, da Universidade de Harvard e do Instituto Max Planck, também publicamos conectomas para porções do cérebro do tentilhão-zebra e das larvas do peixe-zebra .
No trabalho publicado hoje, nossa equipe relata um novo marco: uma reconstrução com resolução sináptica de um pedaço de tecido cerebral humano de 1 mm cúbico. Nossos colaboradores geraram o conjunto de dados usando uma amostra de tecido cerebral do lobo temporal anterior esquerdo que foi removido durante uma cirurgia cerebral em uma pessoa com epilepsia. A equipe de Lichtman usou um microscópio eletrônico de varredura multifeixe para coletar imagens de alta resolução de mais de 5.000 fatias de tecido, cada uma com cerca de 30 nanômetros de espessura. Só a aquisição da imagem levou 326 dias. Em seguida, as ferramentas da nossa equipe costuraram e alinharam os dados da imagem, reconstruíram a estrutura tridimensional de cada célula, incluindo seus axônios e dendritos, identificaram conexões sinápticas e classificaram os tipos de células. A reconstrução revelou várias surpresas.
Desde que publicamos o preprint, expandimos nosso conjunto de ferramentas interativas e de código aberto que permitem aos pesquisadores investigar o conjunto de dados por conta própria. A capacidade de outros pesquisadores revisarem e refinarem esse conectoma do cérebro humano é uma das muitas maneiras pelas quais vemos o lançamento deste artigo e das ferramentas associadas não apenas como o culminar de 10 anos de trabalho, mas como o início de algo novo.
Dimensionando a ciência do cérebro
O primeiro conectoma foi publicado em 1986 — antes da existência de ferramentas de IA — para os 302 neurônios do organismo modelo de nematóides Caenorhabditis elegans . Demorou 16 anos para criá-lo a partir de imagens microscópicas transversais do verme. Os pesquisadores coloriram manualmente as células de uma seção transversal para outra para visualizar as conexões neste sistema nervoso simples.
Quando a equipe Connectomics do Google foi lançada, há dez anos, estávamos entusiasmados com a forma como as inovações em IA e o trabalho com grandes conjuntos de dados poderiam nos permitir passar de 302 neurônios para dezenas de milhares ou milhões encontrados em organismos mais complexos. Nosso trabalho exigiu novos algoritmos capazes de lidar com as enormes quantidades de dados — agora petabytes — que esses estudos geram. Desenvolvemos redes de preenchimento para substituir o esforço manual de colorir células em imagens. Estas redes permitem a reconstrução automatizada de neurônios através de camadas de tecido. Com base nisso, nosso algoritmo SegCLR identifica automaticamente partes distintas de células e tipos de células dentro dessas redes. Também desenvolvemos o TensorStore , uma biblioteca de software C++ e Python de código aberto para armazenar e gerenciar enormes conjuntos de dados multidimensionais. Essa ferramenta obteve benefícios muito além da conectômica e agora é amplamente usada no Google e em toda a comunidade de ML mais ampla.
Demonstramos esses algoritmos pela primeira vez quando lançamos o conectoma para o “hemicérebro” da mosca da fruta Drosophila melanogaster em 2020. Revelando as conexões entre 25.000 neurônios em uma porção central do cérebro da mosca da fruta, esta reconstrução foi usada por outros pesquisadores para fazer descobertas sobre aprendizagem, memória e comportamento na mosca da fruta. Desde então, grupos publicaram centenas de artigos baseados no conectoma da mosca-das-frutas.
Através de colaborações com pesquisadores do Howard Hughes Medical Institute, da Universidade de Harvard e do Instituto Max Planck, também publicamos conectomas para porções do cérebro do tentilhão-zebra e das larvas do peixe-zebra .
No trabalho publicado hoje, nossa equipe relata um novo marco: uma reconstrução com resolução sináptica de um pedaço de tecido cerebral humano de 1 mm cúbico. Nossos colaboradores geraram o conjunto de dados usando uma amostra de tecido cerebral do lobo temporal anterior esquerdo que foi removido durante uma cirurgia cerebral em uma pessoa com epilepsia. A equipe de Lichtman usou um microscópio eletrônico de varredura multifeixe para coletar imagens de alta resolução de mais de 5.000 fatias de tecido, cada uma com cerca de 30 nanômetros de espessura. Só a aquisição da imagem levou 326 dias. Em seguida, as ferramentas da nossa equipe costuraram e alinharam os dados da imagem, reconstruíram a estrutura tridimensional de cada célula, incluindo seus axônios e dendritos, identificaram conexões sinápticas e classificaram os tipos de células. A reconstrução revelou várias surpresas.
Por exemplo, encontramos uma classe de conexões sinápticas raras, mas extremamente poderosas, nas quais um par de neurônios pode estar conectado por mais de 50 sinapses individuais. Enquanto 96,5% dos contatos entre os axônios e suas células-alvo possuem apenas uma sinapse, 0,092% possuem quatro ou mais conexões sinápticas.
A conformação destas ligações combinada com a análise estatística refinada que faz parte da publicação da Science sugere que estas ligações poderosas não são o resultado do acaso, mas sim que estes pares tinham uma razão para estarem mais fortemente ligados do que o normal. Um estudo mais aprofundado dessas conexões poderia revelar seu papel funcional no cérebro. Talvez, por exemplo, estas conexões fortes sejam a forma como o cérebro atinge respostas neurais particularmente rápidas ou como codifica memórias muito importantes.

Também observamos uma curiosa simetria espelhada na camada mais profunda do córtex, que até agora tem sido a camada menos estudada. Esta camada contém os chamados neurônios triangulares que possuem um dendrito basal muito maior que os outros. Entre esses neurônios, 77% caíram em uma de duas categorias: aqueles cujo grande dendrito basal se inclinava em direção a uma borda da amostra e aqueles cujo grande dendrito basal se inclinava em direção à outra borda no ângulo simétrico do espelho. A nossa análise estatística mostrou que aqueles com um tipo de inclinação tendiam a estar próximos de outros com o mesmo tipo de inclinação mais do que seria esperado pelo acaso. A correlação estatística nestes neurônios espelhados sugere que pode haver alguma função subjacente em jogo, mas ainda não sabemos qual poderá ser. Apresentamos aqui um par particularmente simétrico.
A camada mais profunda do córtex contém aglomerados de células que tendem a ocorrer em orientação espelhada umas das outras. Esta imagem mostra um par particularmente simétrico. Crédito: Google Research & Lichtman Lab (Universidade de Harvard). Renderizações de D. Berger (Universidade de Harvard).
Outra estranheza foi o aparecimento de “verticilos de axônios”, que são estruturas lindas, mas misteriosas, nas quais um axônio se envolve em nós complicados. Estes eram raros na amostra e às vezes ocorriam na superfície de outra célula. Da mesma forma, não sabemos o que eles fazem.

Axônios (azul) são a parte filamentosa de uma célula nervosa que transporta um sinal para longe da célula. Os pesquisadores encontraram “redemoinhos de axônios” nunca antes vistos na amostra, às vezes na superfície de outra célula nervosa (amarelo). Crédito: Google Research & Lichtman Lab (Universidade de Harvard). Renderizações de D. Berger (Universidade de Harvard).

O facto de a amostra de tecido cerebral provir de alguém com epilepsia levanta a questão de saber se estas características incomuns estão ligadas à doença ou aos produtos farmacêuticos tomados para tratá-la, em vez de serem comuns aos cérebros humanos. Embora não tenha havido nenhuma indicação substancial de patologia ao observar a amostra com um microscópio óptico, esta possibilidade não pode ser descartada. Repetir esta análise para outras amostras de tecido resultantes do tratamento de outras doenças pode ajudar a contextualizar as descobertas desta amostra.
Dada a escala e complexidade do conjunto de dados, esperamos que existam muitas outras estruturas e características novas ainda a serem descobertas. Essas descobertas são a ponta do iceberg do que esperamos que a conectômica nos diga sobre o cérebro humano.
Ferramentas colaborativas
A ferramenta colaborativa central para interagir com os dados do tecido cerebral é o Neuroglancer , uma plataforma baseada em navegador — originalmente criada para nossa reconstrução do cérebro da mosca da fruta — que permite aos usuários explorar o conjunto de dados, incluindo as anotações geradas por IA do tipo de célula para cada célula.
Outra ferramenta importante é o CAVE , que foi desenvolvido por pesquisadores de Princeton e do Allen Institute como uma extensão do Neuroglancer e permite aos usuários revisar e atualizar os dados anotados para melhorar o conjunto de dados como parte de sua própria pesquisa. Os pesquisadores podem selecionar neurônios específicos ou suas conexões e refinar e anotar manualmente suas reconstruções. CAVE está disponível para conjuntos de dados menores, incluindo o conectoma fly, mas neste trabalho atualizamos os algoritmos para poder lidar com um conjunto de dados deste tamanho. A revisão que esta interface permite é uma etapa final fundamental na conclusão da reconstrução do conectoma.
A nossa esperança é que o novo artigo da Science encoraje outros investigadores a utilizar os dados para fazer descobertas adicionais sobre esta parte do cérebro humano. Estamos satisfeitos que isso já esteja acontecendo. A pré-impressão foi baixada mais de 30.000 vezes e citada em mais de 200 artigos . Isso inclui um estudo recente na Neuron que analisou como pequenos “cílios” ligados às células cerebrais encontradas no conjunto de dados podem desempenhar um papel importante na organização funcional do córtex humano, e uma investigação computacional sobre como a forma dos neurônios se relaciona com outros neurônios. propriedades da organização do córtex humano. O visualizador interativo do conjunto de dados foi acessado mais de 100.000 vezes.


